دستهبندی نشده
آنالیز مرحله سوم (Tertiary Analysis) شامل Annotation
سومین و آخرین مرحله تجزیه و تحلیل NGS به مسأله مهم منطقی سازی داده های مشاهده شده می پردازد. در زمینه ژنتیک انسان ، این روش شامل برقراری ارتباط اساسی بین داده های مختلف و فنوتیپ مشاهده شده در بیمار می باشد.
تجزیه و تحلیل سوم با Variant annotation شروع می شود ، که اطلاعات اضافی بسیار مفید همچون نوع ژن و اطلاعات جمعیتی مربوط به آن را به انواع تشخیص داده شده در مراحل قبلی اضافه می کند. این اطلاعات حاصل از نرم افزارهای پیشگویی کننده عملکرد پروتئین همچون (SIFT, PolyPhen) ، پایگاه های اطلاعاتی مربوط به واریانت ها و بیماری در dbSNP و ClinVarو اطلاعات مربوط به فرکانس جمعیتی در 1000Genome ، COSMIC و PharmGKB می باشد.
DNA annotation یا حاشیه نویسی ژنوم به فرآیند شناسایی مکان ژن ها و مناطق کد کننده در یک ژنوم و تعیین اینکه آن ژن ها چه کاری انجام می دهند گفته می شود. حاشیه نویسی یادداشتی است که از طریق توضیح یا تفسیر اضافه می شود. هنگامی که یک ژنوم توالی یابی شد، برای درک آن باید حاشیه نویسی شود. ژن های موجود در ژنوم یوکاریوتی را می توان با استفاده از ابزارهای حاشیه نویسی مختلف مانند FINDER، حاشیه نویسی کرد.
در طی حاشیه نویسی DNA، یک توالی ناشناخته قبلی با اطلاعاتی همچون موقعیت ژنومی مرزهای اینترون-اگزون، توالی های تنظیمی، تکرارها، نام ژن ها و محصولات پروتئینی غنی می شود.
حاشیه نویسی ژنوم شامل سه مرحله اصلی است:
- شناسایی بخش هایی از ژنوم که پروتئین کد نمی کنند
- شناسایی عناصر روی ژنوم طی فرآیندی به نام پیشبینی ژن
- پیوستن اطلاعات بیولوژیکی به این عناصر
ابزارهای حاشیه نویسی خودکار سعی می کنند این مراحل را برخلاف حاشیه نویسی دستی که نیازمند تخصص انسانی است از طریق تجزیه و تحلیل رایانه ای انجام دهند،. در حالت ایده آل، این رویکردها در یک خط لوله حاشیه نویسی یکسان وجود دارند و یکدیگر را تکمیل می کنند.
یک روش ساده برای حاشیه نویسی ژن با استفاده از ابزارهای جستجو مبتنی بر همسانی، مانند BLAST، برای جستجوی ژن های همولوگ در پایگاه های داده خاص است، سپس اطلاعات به دست آمده برای حاشیه نویسی ژن ها و ژنوم ها استفاده می شود. با این حال، همانطور که اطلاعات به پلت فرم annotation اضافه می شود، حاشیه نویسی های دستی می توانند اختلافات بین ژن هایی را که annotation یکسانی دارند، رفع کنند.
برخی از پایگاههای داده از اطلاعات زمینه ای ژنوم، امتیازهای شباهت، دادههای تجربی و ادغام منابع دیگر برای ارائه حاشیهنویسی ژنوم استفاده میکنند. سایر پایگاههای داده (مانند Ensembl) نیز به منابع دادهای انتخابشده و همچنین طیف وسیعی از ابزارهای نرمافزاری مختلف در پایپ لاین خودکار حاشیهنویسی ژنوم متکی هستند.
حاشیه نویسی ساختاری Structural annotation خود شامل شناسایی عناصر ژنومی از قبیل ORF ها و محلی سازی آنها، ساختار ژن، مناطق کدگذاری، محل قرارگیری توالی های تنظیمی می باشد.
حاشیه نویسی عملکردی Functional annotation شامل پیوست کردن اطلاعات بیولوژیکی همچون عملکرد بیوشیمیایی ، عملکرد بیولوژیکی ،تنظیمات و تعاملات بین ژنی می باشد.
حاشیه نویسی ژنوم همچنان یک چالش بزرگ برای دانشمندانی است که ژنوم انسان را بررسی می کنند، اکنون که توالی ژنوم بیش از هزار انسان (پروژه 100000 ژنوم، انگلستان) و چندین ارگانیسم مدل تا حد زیادی کامل شده است ،دانشمندان هنوز در مراحل اولیه ترسیم قطعات و درک چگونگی انطباق و تعامل این اجزا با هم هستند.
حاشیه نویسی ژنوم یک حوزه تحقیقاتی فعال است و شامل تعدادی سازمان مختلف در جامعه علوم زیستی می شود که نتایج تلاش های خود را در پایگاه های بیولوژیکی در دسترس عموم منتشر می کنند که از طریق وب و سایر وسایل الکترونیکی قابل دسترسی است.
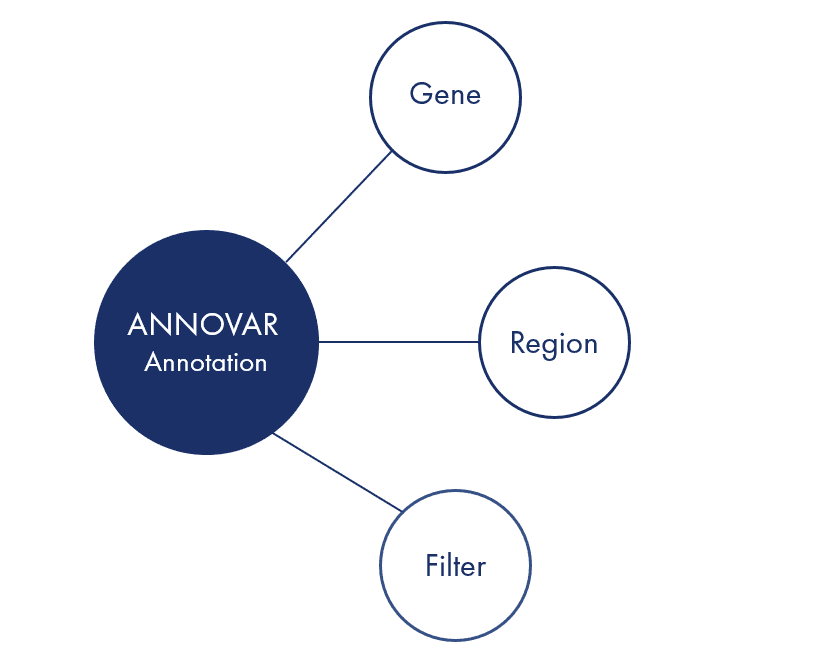
در این میان ANNOVAR یک ابزار نرمافزاری کارآمد برای استفاده از اطلاعات بهروز برای حاشیهنویسی عملکردی انواع ژنتیکی شناساییشده از ژنومهای مختلف (از جمله ژنوم انسانی hg18، hg19، hg38، و همچنین موش، کرم، مگس، مخمر و بسیاری دیگر) می باشد. این ابزار با توجه به لیستی از واریانت ها با شماره کروموزومی، موقعیت شروع، موقعیت پایان، نوکلئوتید مرجع و نوکلئوتیدهای مشاهده شده فعالیت می کند.




من دانشجوی دکترا هستم تزم درباره gwas روی گندم هست. اطلاعات ژنوم من chinese spring v2.1 هست من فایل annotation رو از وب سایت برداشتم ولی نمیدونم چجوری میتونم اطلاعات را مطابقت بدهم
برای تطابق اطلاعات ژنومی با فایل annotation، مراحل زیر را دنبال کنید:
دانلود فایلهای مرتبط:
مطمئن شوید فایلهای زیر را دارید:
فایل ژنوم مرجع (FASTA format)
فایل annotation (GFF3 یا BED format)
نرمافزار تطابقدهی:
از ابزارهای بیوانفورماتیکی مانند BEDTools، SAMtools، یا GATK استفاده کنید. این ابزارها به شما کمک میکنند موقعیتهای ژنتیکی را با اطلاعات annotation تطبیق دهید.
استخراج موقعیتهای مورد نظر:
موقعیت SNPها یا نواحی ژنتیکی مورد نظر را در فایلهای GWAS خود مشخص کنید. معمولاً این اطلاعات در فایل VCF یا جدول CSV/TSV قرار دارد.
تطابق اطلاعات:
از ابزارهایی مانند BEDTools intersect استفاده کنید تا موقعیتهای SNP را با ژنهای مشخص در فایل annotation مقایسه کنید.
میتوانید اطلاعات اضافی مانند نوع ژن، عملکرد، یا مسیرهای زیستی مرتبط را نیز استخراج کنید.
نرمافزارهای بصریسازی:
از نرمافزارهایی مانند IGV (Integrative Genomics Viewer) برای مشاهده تطابق دادهها استفاده کنید.
بررسی پایگاه دادههای مرتبط با گندم:
اگر فایلهای annotation به صورت ناقص هستند یا دقیق نیستند، میتوانید از منابعی مثل Ensembl Plants یا IWGSC اطلاعات بیشتری استخراج کنید.