دستهبندی نشده
رسم شبکه همبیانی (co-expression) با WGCNA
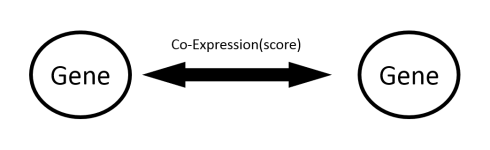
درباره شبکه همبیانی
یکی از مهمترین شبکههای موجود در حوزه سیستم بیولوژی، شبکه همبیانی یا co-expression است. این شبکه حاوی اطلاعاتی درمورد ژنهایی است که بیانشان با یکدیگر بالا و پایین میشود. این موضوع را با طرح یک مساله توضیح میدهیم. فرض کنید میخواهید تفاوت بین دو گروه سالم و بیمار را در بیماری سرطان کلرورکتال بررسی کنید. برای بررسی تفاوت بیانی در ابتدا باید از سایت GEO استفاده کنید که اطلاعاتی در سطوح بیانی دارد.
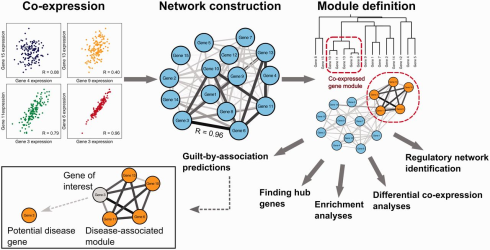
دو مورد از دادههای حاضر در این دیتابیس، دیتای RNA-seq و دیتای میکرواری است. دیتای بیانی به معنای دادهای است که از بیان ژن به دست آمده است. پس برای کشیدن یک شبکه همبیانی یا co-expression شما در ابتدا نیاز دارید اطلاعات خود را از سایت GEO دریافت کنید.
این سایت بخشهای مختلفی دارد. برای به دست آوردن دیتای مدنظر خود در ابتدا باید نام بیماری را در سایت سرچ کنید تا سایت اطلاعات لازم را در قالب GSE یا سریها به شما بدهد. این اطلاعات از مطالعات مختلف جمعآوری شده است. محققان مختلفی از سراسر دنیا برای سوالات زیستی خود از پلتفرمهای مختلفی استفاده کردهاند و دیتای خام و تحلیل شده خود را در قالب سریها وارد سایت NCBI کردهاند.
GEO بخشی از این سایت اصلی است که حاوی این اطلاعات در سطوح بیانی است. پس از سرچ بیماری یا سری مدنظر، شما میتوانید سریها را بر اساس تعداد نمونه یا موارد دیگری که در بخش چپ صفحه نشان داده شده است مرتب کنید. بسته به هدفی که دارید، سری مورد نظر خود را انتخاب میکنید. هرچند ممکن است شما نیاز به دیتای زیادی داشته باشید و به همین دلیل ممکن است از سریهای مختلفی استفاده کنید و دادههای همگی را ترکیب کنید. در این حالت باید به batch effect توجه کنید و تاثیرات آن را از دیتای خود حذف کنید.
پس از این آنکه دیتای موردنظر خود را برای رسم شبکه همبیانی یا co-expression انتخاب کردید، باید به سراغ تحلیل آن بروید. البته در نظر داشته باشید که ممکن است بخشی از کار شما آزمایشگاهی باشد و بجای آن که نیاز داشته باشید از سایتهایی مانند GEO اطلاعات دریافت کنید، خودتان در طرح پژوهشی این اطلاعات آزمایشگاهی را به دست آورده باشید. یعنی بدانید که هر ژن به چه میزانی در نمونههای شما بیان داشته است.
پس از نرمال کردن دیتاست و انجام تحلیلهای آماری برای پیداکردن ژنهای بااهمیت، با تکیه بر مفاهیم آماری و adjusted p value، شما باید ژنهایی را انتخاب کنید که حدآستانه را رد میکنند. یعنی adjusted pvalue زیر 0.05 یا هر اندازهای که در نظر دارید میباشد. این یعنی ژنهای مورد مطالعه از قدرت آماری بالایی برخوردارند و میتوانید ادامه مسیر برای رسم یک شبکه همبیانی یا co-expression را ادامه دهید.
Pvalue آمارهای است که به شما میگوید چقدر احتمال داشته است فرض صفر را به اشتباه رد کرده باشید. فرض صفر در اینجا ادعا میکند که یک ژن خاص تغییر قابل توجهی در مطالعه شما نداشته است. اگر بتوانید آن را رد کنید، شما ژنی دارید که میتوانید به آن اطمینان کنید. پس باید این حدآستانه تا حد امکان پایین باشد و ترجیحا از 0.01 بالاتر نرود. Adjusted p value در نظر دارد که یک ژن را در ارتباط به دیگر ژنهای نمونه بررسی کند. پس متریک قابلاعتمادتری نسبت به pvalue است.
پس آنکه ژنهای خود را بر اساس متریک دیگری به نام fold change نیز فیلتر کردید، تعداد ژنهای اولیه که داشتید کمتر شده است و الان میتوانید به سراغ تحلیلهای بعدی برای رسم شبکه همبیانی یا co-expression بروید. Fold change به شما میگوید که هر ژنی بین نمونه سالم و بیمار چه میزان تغییرات بیانی داشته است. با تکیه بر آن میتوانید بگویید که بیان ژن شما زیاد شده یا کمتر شده است و این تغییر چقدر بوده است.
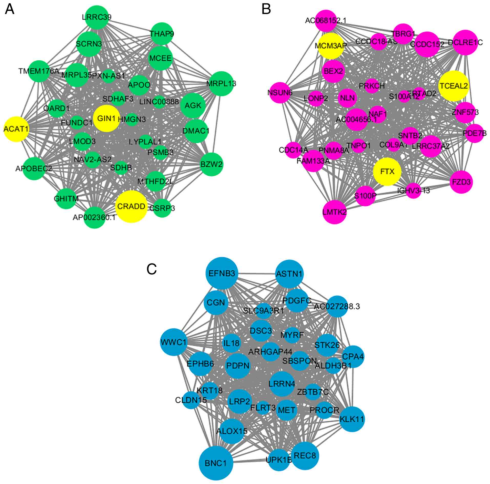
پس از آنکه این فیلترها را انجام دادید، وقت آن میرسد که به کمک پکیج WGCNA یا weighted correlation network analysis که در R و پایتون توسعه داده شده است، شبکه همبیانی یا co-expression را رسم کنید. برای این کار شما باید تعریفی از کورلیشن یا همبستگی داشته باشید. ممکن است فاصله بین ژنی یا distance matrix را به عنوان معیار شباهت در نظر بگیرید یا ترکیبی از روشهای مختلف را در نظر داشته باشید. زمانی که این ماتریس اولیه، یعنی ماتریس شباهت، را ساختید، به کمک دستور adjacency.fromSimilarity میتوانید ماتریس مجاورت را تولید کنید.
این ماتریس حاوی اطلاعاتی از ارتباط نودها با یکدیگر است. نودها در یک شبکه همبیانی یا co-expression، نمایانگر ژنها بوده و اعداد درون جداول، میزان ارتباط بین این نودها را نشان میدهد که همان همبیانی ژنها با یکدیگر است. در این بخش نیز میتوانید یالهایی را نگه دارید که امتیاز بالایی بین نودها دارد.
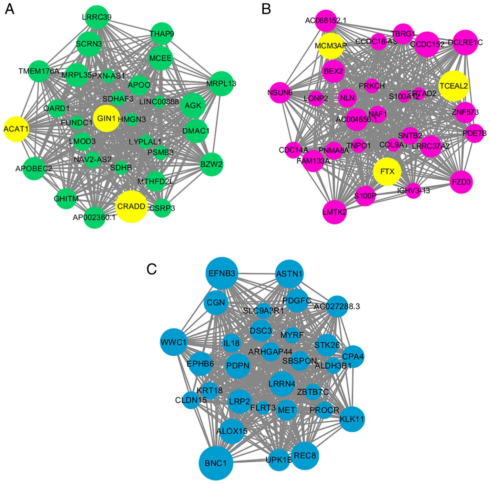
سپس با تبدیل این ماتریس به فرمت قابل خوانش cytoscape یا سایتواسکیپ، میتوانید آن را درون این نرمافزار رسم کنید. برای این کار باید در ستون اول نودهای اولیه یا منبع، ستون دوم نودهای دوم یا مقصد حضور داشته باشند و در ستون سوم نیز مقادیر این ماتریس مجاورت تولید شده توسط پکیچ WGCNA آورده شده باشد.
با ورود این اطلاعات به سایتواسکیپ و مشخص کردن هرکدام برای نرمافزار، میتوانید شبکه همبیانی یا co-expression را رسم کنید. توجه داشته باشید که تعیین حدآستانههایی که در کد اول داشتید، ممکن است ژنهای زیادی را حذف کند. درنتیجه این اتفاق، چون تعدادی از ژنها حذف میشوند، شما نمیتوانید ارتباط بین اجزای شبکه را به درستی مشاهده کنید و با گرافهای جدا افتاده از هم روبهرو خواهید شد. پس سعی کنید حدآستانههایی که تعیین میکنید زیاد از حد سختگیرانه نباشند تا بتوانید نتورک مناسبی داشته باشید.
همچنین شما میتوانید به کمک پکیجهایی مانند iGraph این شبکهها را درون زبان برنامهنویسی پایتون یا R رسم کنید. علاوه بر این موارد، با در دست داشتن ژنهای مورد مطالعه میتوانید از سایتهایی مانند GeneMania استفاده کنید تا شبکه همبیانی یا co-expression را با تکیه بر دیتابیس خود بسازد. ممکن است با تحلیل این شبکهها ژنهایی را پیدا کنید که با کنترل بیان تعدادی از ژنها، بیماری را القا میکند.