تکنیک ها
نرم افزار Autodock
اتوداک یک برنامه شبیه ساز مولکولی است و به خصوص برای اتصال لیگاند به پروتئین کاربرد دارد. امروزه اتوداک 4 در دسترس عموم قرار گرفته اما نسخه بهبود یافته آن اتوداک ویناVina است.
در حال حاضر AutoDock و Vina توسط Scripps Research ، به ویژه مرکز زیست شناسی ساختاری محاسباتی به رهبری دکتر آرتور جی اولسون نگهداری می شوند.
AutoDock از دو برنامه اصلی تشکیل شده است: AutoDock برای اتصال لیگاند به مجموعه ای از شبکه ها که پروتئین هدف را توصیف می کنند. AutoGrid برای پیش محاسبه این شبکه ها. استفاده از AutoDock به کشف چندین دارو از جمله مهارکننده های HIV1 اینتگراز کمک کرده است.
اگر به سایت اتوداک سر بزنید خود سایت اتوداک وینا را پیشنهاد میدهد چراکه نسخه وینا از بسیار جهات از اتوداک معمولی بهتر و سرعت بالاتری دارد.
در اتوداک هدف داک کردن لیگاند به رسپتور است از این سو یک ماده رسپتور یا گیرنده است و ماده دیگر لیگاند یا متصل شونده است از آنجایی که در اتصال دوماده بهم نقاط خاصی از ماده در اتصال بهم نقش دارند در نرم افزار اتوداک ایجاد گرید باکس نقش ویژه ای داشته و باید محل فعال هر رسپتور توسط nbci شناسایی شود و آن قسمت توسط گرید باکس مشخص شود
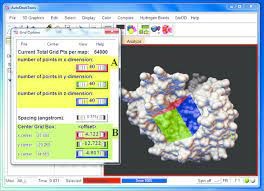
در شکل زیر نمایی از محیط اتوداک و گرید باکس نمایش داده شده است.
همان طور که در شکل میبینید یک پروتئین در این برنامه توسط یک گرید باکس محاصره شده است درواقع گرید باکس همان ناحیه ای است که ما مایل هستیم لیگاند ما متصل شود بعد از آنجام فرایند داک لازمه بفهمیم آیا واقها داک صورت گرفته یا خیر بدین منظور یک روش کیفی و یک روش کمی وجود دارد در روش کیفی تصاویری از داک نشان داده میشود که دقیقا میتوان دید که لیگاند به کدام قسمت متصل شده، اما این پاسخی قاطع نخواهد بود برای پاسخ قاطع نیاز به برسی کمی داریم در برسی کمی بعد داک مقدار انرژی ازاد واکنش داده میشود که منفی تر بودن انرژی آزاد گیبس دال بر اتصال موفق لیگاند به رسپتور است برای اطمینان بیشتر سایتی تحت عنوان swiss dock وجود دارد که در عرض چند دقیقه میتونه فرایند داک را به طور خودکار انجام دهد و نتاج انرژی آزاد گیبس داده میشود. در نتیجه میتوان نتایج را با نتایج این سایت مقایسه کرد.لازم به ذکر است سایت معرفی شده تنها کاربرد برای مقایسه داشته و نمیتوان از آن برای کار های تحقیقاتی استفاده کرد.