اطلاعات عمومی,ویکی ژن
ساختار Stem-Loop (ساختار سنجاقسری): ویژگیها، انواع، نمونه و کاربردها
ساختار Stem-Loop
مولکول های DNA معمولاً به شکل دو رشتهای و در مقابل RNA به شکل تک رشتهای وجود دارد. با این حال، هم DNA و هم RNA در فرم تک رشتهای ممکن است ساختار حلقه مانندی به نام ساختار Stem-Loop یا سنجاق سر ایجاد کنند. حلقه سنجاقسری (تابخوردگی ساختاری ثانویه) هم در اسید ریبونوکلئیک (RNA) و هم در اسید دئوکسی ریبونوکلئیک (DNA) مشاهده میشود، اما این پدیده بیشتر در RNA رخ میدهد.

ساختار Stem-Loop (ساختار سنجاقسری) چیست؟
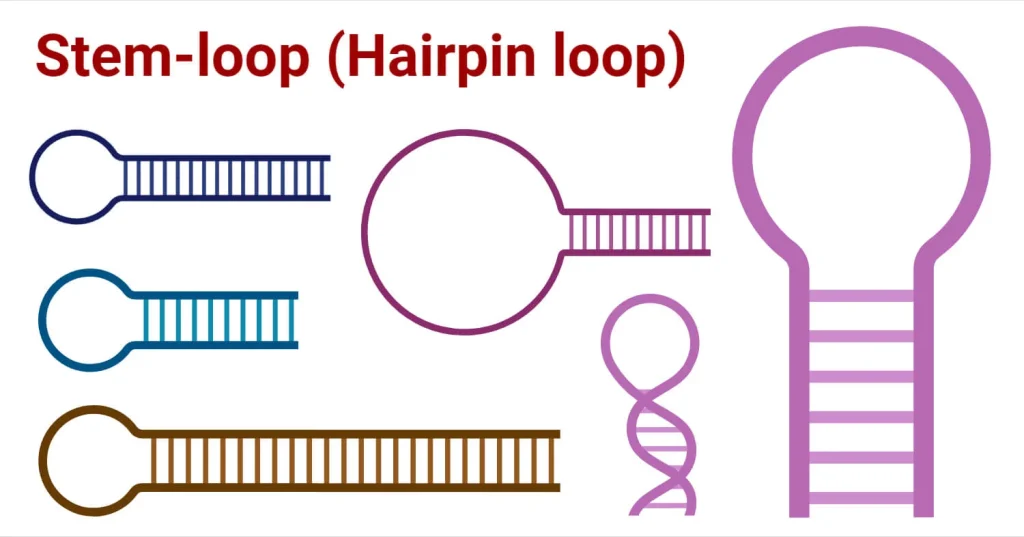
ساختار Stem-Loop را میتوان به عنوان یک الگوی سنجاق سر تعریف کرد که در اثر جفت شدن بازهای توالی نوکلئوتیدی (به ویژه در توالی پالیندرومیک) در همان رشته ایجاد شده است. در واقع، نوکلئوتیدها در داخل مولکول جفت میشوند تا یک ساختار مارپیچ دو رشتهای را تشکیل دهند که انتهای آن به یک حلقه فاقد جفت باز ختم میشود و در نتیجه ساختاری شبیه سنجاق سر ایجاد میکند.
ویژگیهای ساختار Stem-Loop (ساختار سنجاقسری)
- حلقه های سنجاقسری در اسیدهای نوکلئیک تک رشتهای تشکیل میشوند و شامل یک ساقه متشکل از جفت باز و یک توالی حلقوی فاقد جفت باز میباشند.
- تعداد نوکلئوتیدهای جفت نشده داخل حلقه از 7 تا 20 جفت باز میتواند متغیر باشد.
- اغلب برای توصیف آنها از تعداد بازهای جفت نشده در حلقه استفاده میشود بدین ترتیب که پیشوندهایی چون tri-، tetra-، penta- را کنار اسم loop (حلقه) میآورند.
- ساختار سنجاق سر نواحی مکملی را در همان تک رشته جفت باز تشکیل میدهند و یک مارپیچ دو رشتهای را ایجاد میکنند که به یک حلقه فاقد بازهای جفت شده ختم میشود. ساختار آنها در اسید نوکلئیک شامل یک ساقه دارای بازهای جفت شده و یک توالی حلقوی با نوکلئوتیدهای جفت نشده یا جفت شده مدل غیر‑واتسون‑ کریک میباشد. ساختار نهایی شبیه یک حلقه یا U شکل است.
- در RNA، مولکولهای mRNA میتوانند حلقههای سنجاق سر را زمانی که دو توالی مکمل پس از تا خوردگی به یکدیگر متصل میشوند، تشکیل دهند.
- مشخص شده است که حلقههای سنجاق سر با موتیف سه نوکلئوتیدی GNA مرکزی (G، گوانین؛ N، هر نوکلئوتید؛ A، آدنین) ساختارهای پایداری را تشکیل میدهند. به عنوان مثال، دمای ذوب گذار جهت اختلال در ساختار سنجاقسر برای توالی 5ꞌ‑GCGCAGC، 67 درجه سانتیگراد گزارش شده است.
توجه: تشکیل ساختار سنجاقسری در DNA نیز رخ میدهد که بسیار سریع ، معمولاً در عرض میکروثانیه، فراتر از حداکثر مقیاسهای زمانی شبیهسازی دینامیک مولکولی فعلی روی میدهد.

منشا سنجاق سر RNA
سنجاق سر RNA شامل یک ساقه RNA دو رشته ای (dsRNA) با عدم تطابق و برآمدگی، همراه با یک حلقه انتهایی است که در پیشبینیهای تا خوردگیهای RNA، یک ساختار ثانویه رایج است.
سنجاق سر RNA از طریق دو مکانیسم شکل میگیرد:
- رونویسی از توالیهای تکراری معکوس DNA توسط RNA پلیمراز وابسته به DNA، که منجر به تا خوردگی RNA و تشکیل ساختار سنجاقسر میشود.
- تشکیل مولکول RNA به عنوان یک الگو برای RNA پلیمراز وابسته به RNA که منجر به سنتز ساقه دوم رشته میشود. این مکانیسم، که عمدتاً در فرآیندهای خاص تکثیر ویروسی مشاهده میشود، ساختارهای سنجاقسری بلند و کامل dsRNA را تولید میکند. مکانیسم تولید این dsRNAها اغلب از نوع مکانیسم تکثیر «copy-back» است.
توجه: RNA سنجاقسر، یک المان تنظمی بیان ژن سیس و ترانس است:
- در سیس: RNAهای سنجاقسر مولکول RNA را در جایی که قرار دارند تنظیم میکنند.
- در ترانس: RNAهای سنجاقسر بر سایر RNA ها یا بر مسیرهای متابولیکی خارج از منشا خود تأثیر میگذارند.
مولکولهایی با ساختار حلقوی:
- حلقه‑ ساقهها اجزای جدایی ناپذیر ساختارهای RNA مانند pre-microRNA، RNA انتقالی (tRNA)، شبهگرههای RNA (RNA pseudoknots) و ریبوزیمها هستند.
- در tRNA، آنتی کدون شناسایی کننده کدون روی یک حلقه جفت نشده قرار دارد، در حالی که شبهگرههای RNA دارای ساختارهای Stem-Loop تو در تو (nested stem-loop) هستند.
- ریبوزیم هایی مانند ریبوزیم سر چکشی نیازمند ساختارهای حلقه-ساقه هستند که سه ساختار Stem-Loop فعالیت خود شکافی را تسهیل میکنند.
- حلقههای سنجاق سر درون 5ꞌUTR پروکاریوتی اغلب ترجمه را تنظیم میکنند یا با پروتئینهای تنظیمکننده تعامل دارند.
عوامل پایداری stem-loop (حلقه سنجاق سر)
- پایداری ساختارهای Stem-Loop به پایداری نواحی مارپیچ و حلقه آنها بستگی دارد که توسط عوامل مختلف کنترل میشود.
- پایداری جفت بازها تحت تأثیر توالی، طول، عدم تطابق (mismatches) و نیز ترکیب باز میباشد، به طوری که جفت بازهای گوانین-سیتوزین پایدارتر از جفت بازهای آدنین-اوراسیل هستند.
- RNA معمولاً دارای جفتهای گوانین-اوراسیل است، در حالی که برهمکنشهای انباشتگی بازها (base stacking interactions) به پایداری مارپیچ کمک میکنند.
- پایداری حلقه بسیار مهم است. تشکیل حلقههای کوتاهتر از سه باز غیرممکن هستند. حلقههای بزرگ و فاقد ساختار ناپایدار هستند.
- طول بهینه حلقه معمولاً از 4 تا 8 باز متغیر است. توالیهایی مانند UUCG تترالوپ های پایداری را ایجاد کرده و تشکیل سریع را تسهیل میکنند.
نمونه هایی از Stem-Loop (حلقه سنجاق سری)
- حلقههای سنجاق سری پدیده رایج در رشتههای اسید نوکلئیک هستند که در موارد مختلفی رخ میدهند.
- در پروکاریوتها، یکی از نمونههای حلقه سنجاق سری، توالی پایان برای رونویسی است، جایی که مواجهه با این حلقه باعث جدا شدن پلیمراز و در نتیجه پایان رونویسی میشود.
- مثال قابل توجه دیگر tRNA، جزء کلیدی در سنتز پروتئین، است که دارای ساختار حلقههای
سنجاق سری میباشد. - به طور خاص، مولکول tRNA دارای سه حلقه سنجاق سری شبیه شبدر سه برگ است. یکی از این 3 حلقه حاوی یک توالی مهم به نام آنتی کدون است که مسئول شناسایی و رمزگشایی مولکولهای mRNA در طول ترجمه است.
- این تشکل برگ شبدر پیوند دقیق بین کدونها، آنتی کدونها و اسیدهای آمینه مربوطه را تسهیل میکند، امری که برای سنتز دقیق پروتئین ضروری میباشد.
توالی پالیندرومیک —CCTGCXXXXXXXGCAGG— میتواند ساختار سنجاق سری زیر را ایجاد کند:
—C G—
C G
T A
G C
C G
X X
X X
X X
X
مثال بلندتری از توالی RNA 5ꞌ به 3ꞌ که میتواند منجر به ساختار حلقه سنجاقسری شود به شرح زیر است:
GCCGCGGGCCGAAAAAACCCCCCCGGCCCGCGGC
اهمیت Stem-Loop (حلقه سنجاق سری)
-
اهمیت ساختار Stem-Loop RNA:
- ساختارهای Stem-Loop RNA برای اندازهگیریهای در-لحظه و ساخت حسگرهای زیستی ارزشمند هستند، زیرا پروبهای الیگونوکلئوتیدی با ساختار Stem-Loop که به درستی طراحی شده باشند، میتوانند در حضور توالیهای مکمل باز شوند و امکان شناسایی اهداف بیومولکول علامت گذاری نشده مانند اسیدهای نوکلئیک و پروتئینها را فراهم کنند.
- در پروکاریوتها، در پایان رونویسی مستقل از rho، تشکیل حلقه سنجاق سری در mRNA باعث جدا شدن RNA پلیمراز از الگوی DNA میشود.
- ساختارهای Stem-Loop RNA به عنوان محل اتصال برای پروتئینهای مختلف عمل میکنند.
- ساختارهای Stem-Loop RNA به عنوان سوبسترا برای واکنشهای آنزیمی عمل میکنند.
- ساختارهای Stem-Loop RNA فعالیتهای آنزیمی ذاتی (intrinsic) را نشان میدهند.
- ساختارهای کوتاه سنجاق سری در mRNA در فرآیندهای متنوعی مانند استقرارسازی mRNA، تنظیم ترجمه و حفظ ثبات دخیل است.
- ساختارهای Stem-Loop از طریق محلهای ورودی ریبوزومی داخلی به شروع ترجمه کمک میکنند و در تکثیر ویروس نقش دارند.
- در مولکولهای RNA، حلقههای سنجاقسری بهعنوان جایگاههای هستهسازی برای تاخوردگی RNA عمل میکنند و بدین ترتیب بر تشخیص پروتئین RNA و تنظیم ژن تأثیر میگذارند.
- ساختارهای Stem-Loop، مانند فانوسهای مولکولی عمل میکنند که تشخیص دقیق توالی هدف را ممکن میسازد و فرصتی برای ایجاد و توسعه حسگرهای تشخیصی نوآورانه و روشهای درمانی فراهم میکنند.
-
اهمیت ساختار سنجاق سری DNA:
- فاژهای تک رشتهای به طور گسترده از ساختار سنجاق سری DNA در طول چرخه زندگی خود، از جمله در مبدا تکثیر coli، استفاده میکنند.
- DNA صلیبی، دارای موتیف های سنجاق سری، در مکان های ژنومی مختلف، مانند پروموترهای فاژ RCR dso و N4 مشاهده شده است.
- در یوکاریوتها، پروتئینهایی کشف شدهاند که به DNA صلیبی متصل میشوند. این امر حاکی از دخالت آنها در انتقال ژنوم و شروع تکثیر است.
- زیستشناسی مولکولی و بیوتکنولوژی: پروبهای الیگومر سنجاق سری DNA، ابزاری ضروری در بیوتکنولوژی و تشخیص هستند چرا که این پروبها اختصاصیت بالایی در هیبریداسیون پروب/هدف دارند و بدین ترتیب میتوانند به تمایز تطابق در مقابل عدم تطابق کمک کنند.
همچنین بخوانید:
مترجم: الهام مظاهری