اطلاعات عمومی
آنزیم محدودکننده چیست؟
آنزیم محدودکننده
آنزیمهای محدودکننده، اندونوکلئاز محدودکننده یا به اصطلاح محدودکنندهها، آنزیمهایی هستند که DNA را به قطعاتی در داخل یا نزدیک مکانهای شناسایی خاص درون مولکولهایی که به عنوان مکانهای محدود شده شناخته میشوند، میشکنند. آنزیمهای محدودکننده تنها یک دسته از گروه وسیعتر آنزیمهای اندونوکلئاز هستند. این آنزیمها معمولاً به پنج نوع طبقهبندی میشوند که از نظر ساختار و اینکه آیا قادر به برش سوبسترای DNA خود در محل تشخیص خود هستند یا مکانهای تشخیص و برش از یکدیگر جدا میباشند، متفاوتند. به منظور برش DNA، تمام آنزیمهای محدودکننده دو برش ایجاد میکنند که یکی از آنها از طریق ستون قند-فسفات (یعنی هر رشته) مارپیچ دوگانه DNA است. این آنزیمها در باکتریها و آرکیها یافت شده و یک مکانیسم دفاعی را در برابر ویروسهای مهاجم ایجاد میکنند. در داخل یک پروکاریوت، آنزیمهای محدودکننده به صورت انتخابی، DNA خارجی را طی فرآیندی به نام هضم محدود، برش میدهند. در همین حال، DNA میزبان توسط یک آنزیم اصلاح کننده (متیل ترانسفراز) محافظت میشود که DNA پروکاریوتی را تغییر داده و از برش جلوگیری میکند. این دو فرآیند با هم سامانه اصلاح محدودیت را تشکیل میدهند.
بیش از ۳۶۰۰ اندونوکلئاز محدودکننده شناخته شده است که بیش از ۲۵۰ ویژگی مختلف دارند. بیش از ۳۰۰۰ مورد از این موارد به تفصیل مورد مطالعه قرار گرفتهاند و بیش از ۸۰۰ مورد از آنها به صورت تجاری در دسترس هستند. این آنزیمها به طور معمول برای اصلاح DNA در آزمایشگاهها استفاده میشوند و ابزاری حیاتی در شبیه سازی مولکولی (کلونینگ) هستند.
تاریخچه
اصطلاح آنزیم محدودکننده از مطالعات فاژ λ (ویروسی که باکتریها را آلوده میسازد) و پدیده محدودیت کنترل شده توسط میزبان و اصلاح چنین فاژ یا باکتریوفاژ باکتریایی نشات گرفته است. این پدیده اولین بار در آزمایشگاههای سالوادور لوریا، ژان وایگل و جوزپه برتانی (Salvador Luria, Jean Weigle and Giuseppe Bertani) در اوایل دهه ۱۹۵۰ شناسایی شد. پس از مدتی مشخص گردید که باکتریوفاژ λ (میتواند در یک سویه از اشریشیا کلی به خوبی رشد کند) مانند E. coli C هنگامی که در سویه دیگری، به عنوان مثال E. coli K رشد کند، بازده آن میتواند به میزان قابل توجهی (۳ تا ۵ مرتبه) کاهش یابد. سلول میزبان، در این مثال E. coli K، به عنوان میزبان محدودکننده شناخته شده و به نظر میرسد که توانایی کاهش فعالیت بیولوژیکی فاژ λ را دارد. اگر فاژی در یک سویه مستقر شود، توانایی آن فاژ برای رشد در سویههای دیگر نیز محدود میگردد. در دهه ۱۹۶۰، طی پژوهش انجام گرفته در آزمایشگاههای ورنر آربر و متیو مزلسون (Werner Arber and Matthew Meselson) گزارش شد که این محدودیت ناشی از برش آنزیمی DNA فاژ است و بنابراین آنزیم درگیر، آنزیم محدودکننده نامیده میشود.
آنزیمهای محدودکننده مورد مطالعه توسط Arber و Meselson آنزیمهای محدود کننده نوع I بودند که DNA را به طور تصادفی از محل تشخیص جدا میکنند. در سال ۱۹۷۰، Hamilton O. Smith, Thomas Kelly و Kent Wilcox اولین آنزیم محدودکننده نوع II، به نام HindII را از باکتری هموفیلوس آنفولانزا جدا و شناسایی کردند. این نوع از آنزیمهای محدودکننده برای پژوهشهای آزمایشگاهی مفیدتر هستند زیرا DNA را در محل توالی شناسایی خود میشکافند و بیشترین استفاده را به عنوان ابزار زیستشناسی مولکولی دارند. پس از آن، Daniel Nathans و Kathleen Danna showedنشان دادند که برش DNA ویروس سیمیان ۴۰ (SV40) توسط آنزیمهای محدودکننده، قطعات خاصی را ایجاد میکند که میتوان آنها را با استفاده از الکتروفورز ژل پلی آکریل آمید جدا نمود، بنابراین نتیجه شد که از آنزیمهای محدودکننده نیز میتوان برای نقشهبرداری DNA (Mapping) استفاده کرد. جایزه نوبل فیزیولوژی یا پزشکی در سال ۱۹۷۸ به دلیل کشف و شناسایی آنزیمهای محدودکننده به Werner Arber, Daniel Nathans, and Hamilton O. Smith تعلق گرفت. کشف آنزیمهای محدودکننده، امکان دستکاری و تغییر DNA را فراهم میکند و منجر به توسعه فناوری DNA نوترکیب شده که کاربردهای زیادی دارد، به عنوان مثال، اجازه میدهد تا در مقیاس وسیع پروتئینهایی مانند انسولین انسانی مورد استفاده بیماران دیابتی را تولید کند.
ریشه
آنزیمهای محدودکننده احتمالاً از یک نیای مشترک تکامل یافته و از طریق انتقال افقی ژن گسترده شدهاند. علاوه بر این، شواهد فزایندهای نیز وجود دارد که نشان میدهد اندونوکلئازهای محدودکننده به عنوان یک عنصر ژنتیکی خودخواه تکامل یافتهاند.
جایگاه شناسایی
آنزیمهای محدودکننده، توالی خاصی از نوکلئوتیدها را شناسایی نموده و یک برش دو رشتهای در DNA ایجاد میکنند. توالیهای شناسایی را میتوان بر اساس تعداد بازها در محل تشخیص آن، معمولاً بین ۴ تا ۸ باز طبقهبندی کرد و تعداد بازها در توالی تعیین میکند که هر چند وقت یکبار جایگاه بهطور تصادفی در هر ژنوم مشخص ظاهر میشود، به عنوان مثال، توالی جفت ۴ باز از نظر تئوری یک بار در هر ۴^۴ یا ۲۵۶ جفت باز، ۶ باز، ۶^۴ یا ۴۰۹۶ جفت باز و ۸ باز یا ۸^۴ یا ۶۵۵۳۶ جفت باز خواهد بود. بسیاری از آنها توالیهای پالیندرومیک هستند؛ به این معنی که توالیهای باز، از هر دو سو به یک صورت خوانده میشوند. در حالت تئوری، دو نوع توالی پالیندرومی وجود دارد که میتواند در DNA امکان پذیر باشد. حالت پالیندروم آینهمانند شبیه به نمونههایی است که در متن معمولی یافت میشود و در آن یک دنباله روی رشته DNA از هر دو سمت به یک شکل خوانده میشود، مانند GTAATG.
حالت پالیندروم تکرار معکوس نیز توالی است که به صورت یکسان به جلو و عقب میخواند، اما توالیهای رو به جلو و عقب در رشتههای DNA مکمل (یعنی DNA دو رشتهای) یافت میشوند، مانند GTATAC (GTATAC مکمل CATATG است). پالیندرومهای تکرار معکوس رایجترند و اهمیت بیولوژیکی بیشتری نسبت به پالیندرومهای آینه مانند دارند.
برش EcoRI انتهای “چسبنده” تولید میکند:
در حالی که برش آنزیم محدودکننده SmaI انتهای “بلند” ایجاد میکند:
توالیهای تشخیص در DNA برای هر آنزیم محدودکننده متفاوت است، که باعث ایجاد تفاوت در طول، توالی و جهت رشته (انتهای ‘۵ یا انتهای ‘۳) یک انتهای چسبنده حاصل از برش آنزیمی میشود.
آنزیمهای محدودکننده مختلف که توالی یکسانی را تشخیص میدهند به عنوان نئوشیزومر شناخته میشوند. اینها اغلب در نواحی مختلف توالی جدا میشوند. آنزیمهای مختلفی که در یک مکان تشخیص داده و میشکنند، ایزوشیزومر نامیده میشوند.
انواع
اندونوکلئازهای محدودکننده طبیعی بر اساس ترکیب و نیازهای کوفاکتور آنزیمی، ماهیت توالی هدف و موقعیت محل برش DNA آنها نسبت به توالی هدف به چهار گروه (نوع I، II III و IV) دسته بندی میشوند. با این حال، آنالیز توالی DNA آنزیمهای محدودکننده، تغییرات زیادی را نشان میدهد که این خود نشانه وجود بیش از چهار نوع میباشد. همه انواع آنزیمها، توالیهای کوتاه DNA خاصی را شناسایی کرده و برش اندونوکلئولیتیک DNA را انجام میدهند تا قطعات خاصی با ‘۵-فسفاتهای پایانی به دست آید. همانطور که در زیر خلاصه میگردد: آنها در توالی تشخیص، ترکیب زیر واحد، موقعیت برش و نیازهای کوفاکتور متفاوت هستند:
- آنزیمهای نوع I (EC 3.1.21.3) در مکانهای دور از یک مکان شناسایی را میشکنند. برای عملکرد به هر دو ATP و S-adenosyl-L-methionine نیاز دارند. پروتئین چند منظوره با فعالیتهای برش محدود و متیلاز (EC 2.1.1.72).
- آنزیمهای نوع II (EC 3.1.21.4) در داخل یا در فواصل خاص کوتاه از یک محل تشخیص را برش میدهند. بیشتر آنها به منیزیم نیاز دارند. آنزیمهای تک عملکردی (برش محدود) مستقل از متیلاز.
- آنزیمهای نوع III (EC 3.1.21.5) در مکانهایی در فاصله کوتاهی از یک مکان شناسایی را برش میدهند. به ATP نیاز دارند (اما آن را هیدرولیز نمیکنند). S-adenosyl-L-methionine واکنش را تحریک میکند اما لازم نیست؛ به عنوان بخشی از یک مجموعه با متیلاز اصلاح شده وجود دارد (EC 2.1.1.72).
- آنزیم های نوع IV ، DNA تغییر یافته مانند DNA متیله، هیدروکسی متیله و گلوکوزیل هیدروکسی متیله شده را هدف قرار میدهند.
- آنزیم های نوع V از RNAهای راهنما (gRNA) استفاده میکنند.
نوع I
آنزیمهای محدود کننده نوع I اولین آنزیمهایی بودند که برای اولین بار در دو سویه مختلف (K-12 و B) E. coli شناسایی شدند این آنزیمها در محلی متفاوت و با فاصله تصادفی (حداقل ۱۰۰۰ جفت باز) از محل تشخیصشان برش انجام میدهند. شکاف در چنین مکانهای تصادفی به دنبال فرآیند جابهجایی DNA است که نشان میدهد این آنزیمها نیز موتورهای مولکولی هستند. محل تشخیص نامتقارن است و از دو بخش خاص تشکیل شده است (یکی شامل ۴-۳ نوکلئوتید و دیگری حاوی ۵-۴ نوکلئوتید) که توسط یک فاصلهگذار غیر اختصاصی حدود ۸-۶ نوکلئوتید از هم جدا شدهاند. این آنزیمها چند منظوره هستند و بسته به وضعیت متیلاسیون DNA هدف، قادر به هضم محدود و فعالیتهای مودیفیکاسیون یا دستکاری و اصلاح هستند. کوفاکتورهای S-Adenosyl methionine (AdoMet)، آدنوزینتریفسفات هیدرولیز شده (ATP) و یونهای منیزیم (Mg+2) برای فعالیت کامل آنها مورد نیاز میباشند. آنزیمهای محدودکننده نوع I دارای سه زیر واحد به نام های HsdR، HsdM و HsdS هستند. HsdR برای برش محدودکننده مورد نیاز است. HsdM برای افزودن گروههای متیل به DNA میزبان (فعالیت متیل ترانسفراز) ضروری است، HsdS علاوه بر فعالیت هضم محدود (برش DNA) و اصلاح و تغییر (DNA متیل ترانسفراز)، برای اختصاصی بودن مکان شناسایی (اتصال به DNA) نیز مهم است.
نوع II
آنزیمهای محدودکننده نوع دوم از چندین جهت با آنزیمهای محدودکننده نوع I متفاوت هستند. آنها همودایمرهایی با مکانهای تشخیصی که معمولاً تقسیم نشده و پالیندروم هستند و طول آن ۴ تا ۸ نوکلئوتید است را تشکیل میدهند. این آنزیمها DNA را در همان محل شناسایی کرده و میشکنند و از ATP یا AdoMet برای فعالیت خود استفاده نمیکنند، آنها معمولاً فقط به Mg+2 به عنوان کوفاکتور نیاز دارند. این آنزیمها پیوند فسفودیاستر DNA مارپیچ دوگانه را برش میدهند و یا میتوانند مرکز هر دو رشته را شکاف دهند تا انتهایی صاف ایجاد کند یا در یک موقعیت ناپایدار، برآمدگیهایی به نام انتهای چسبناک ایجاد نماید. در دهه ۱۹۹۰ و اوایل دهه ۲۰۰۰، آنزیمهای جدیدی از همین خانواده کشف شد که از تمام معیارهای کلاسیک این کلاس آنزیمی پیروی نمیکرد و نامگذاری زیرخانوادههای آنها بر اساس انحراف از ویژگیهای معمول آنزیمهای نوع II ایجاد گردید. این زیرگروهها با استفاده از پسوند حرف تعریف میشوند. آنزیمهای محدودکننده نوع IIB (به عنوان مثال، BcgI و BplI) مولتیمرهایی هستند که حاوی بیش از یک زیر واحد هستند. آنها DNA را از دو طرف تشخیص داده و از همان محل میشکافند و همچنین به هر دو کوفاکتور AdoMet و Mg+2 نیاز دارند. اندونوکلئازهای محدودکننده نوع IIE (به عنوان مثال NaeI) پس از برهمکنش با دو نسخه از توالی شناسایی خود، DNA را میبرند. محل تشخیص به عنوان هدف برش عمل میکند، در حالی که دیگری به عنوان یک افکتور آلوستریک عمل میکند که کارایی برش آنزیم را تسریع نموده یا بهبود میبخشد. مشابه آنزیمهای نوع IIE، اندونوکلئازهای محدودکننده نوع IIF (مانند NgoMIV) با دو نسخه از توالی تشخیص خود برهمکنش دارند اما هر دو توالی را همزمان میشکافند. اندونوکلئازهای محدودکننده نوع IIG (مانند RM.Eco57I) مثل آنزیمهای محدودکننده کلاسیک نوع II دارای یک زیر واحد هستند، اما برای فعال بودن به کوفاکتور AdoMet نیاز دارند. اندونوکلئازهای محدودکننده نوع IIM مانند DpnI، قادر به شناسایی و برش DNA متیله هستند. اندونوکلئازهای محدودکننده نوع IIS (به عنوان مثال، FokI) DNA را در فاصله مشخصی از مکانهای تشخیص نامتقارن غیر پالیندرومیک خود برش میدهند. این ویژگی به طور گستردهای برای انجام تکنیکهای شبیهسازی in vitro مانند شبیهسازی گلدن گیت استفاده میشود. این آنزیمها ممکن است به عنوان دایمر عمل کنند. به طور مشابه، آنزیمهای محدودکننده نوع IIT (به عنوان مثال، Bpu10I و BslI) از دو زیر واحد مختلف تشکیل شدهاند. برخی توالیهای پالیندرومیک را تشخیص میدهند در حالی که برخی دیگر مکانهای شناسایی نامتقارن دارند.
نوع III
آنزیمهای محدودکننده نوع III (به عنوان مثال، EcoP15) دو توالی غیرپالیندرومیک جداگانه را شناسایی میکنند که جهتگیری معکوس دارند. آنها DNA را حدود ۲۰ تا ۳۰ جفت باز پس از محل تشخیص برش میدهند. این آنزیمها حاوی بیش از یک زیرواحد هستند و به ترتیب به کوفاکتورهای AdoMet و ATP برای نقش خود در متیلاسیون DNA و هضم محدود نیاز دارند. آنها اجزایی از مکانیسمهای محدودکننده-اصلاح DNA پروکاریوتی هستند که از ارگانیسم در برابر مهاجمین خارجی DNA محافظت میکنند. آنزیمهای نوع III، پروتئینهای هتروالیگومری و چند عملکردی هستند که از دو زیرواحد Res (P08764) و Mod (P08763) تشکیل شدهاند. زیرواحد Mod، توالی خاصی از DNA سامانه را تشخیص میدهد و یک متیل ترانسفراز اصلاحکننده است. بدین ترتیب، از نظر عملکردی معادل زیرواحدهای M و S اندونوکلئاز محدودکننده نوع I است. Res برای برش مورد نیاز است، اگرچه به خودی خود فعالیت آنزیمی ندارد. آنزیمهای نوع III، توالیهای DNA نامتقارن کوتاه ۶-۵ جفت باز را شناسایی کرده و ۲۷-۲۵ جفت باز در پایین دست را برش میدهند تا برآمدگیهای کوتاه و تک رشته ای ‘۵ را بر جای بگذارند. آنها جهت هضم محدود، نیاز به حضور دو محل تشخیص غیرمتیله معکوس دارند. این آنزیمها تنها یک رشته از DNA را در موقعیت ۶N- باقیماندههای آدنوزیل، متیله میکنند بنابراین DNA تازه تکثیر شده تنها یک رشته متیله خواهد داشت که برای محافظت در برابر هضم محدود کافی است. آنزیمهای نوع III متعلق به زیرخانواده بتا ۶N آدنین متیل ترانسفرازها هستند که شامل ۹ موتیف است که مشخصه اصلی این خانواده میباشد: از جمله موتیف I، محفظه اتصال AdoMet (FXGXG) و موتیف IV، ناحیه کاتالیزوری (S/D/N (PP) Y/F).
نوع IV
آنزیمهای نوعIV معمولاً DNA متیلهشده و تغییر یافته را شناسایی میکنند، سیستمهای از نوع McrBC و Mrr در E. coli مثالهایی از این نوع هستند.
نوع V
آنزیمهای محدودکننده نوع V (به عنوان مثال، کمپلکس cas9-gRNA از CRISPRs) از RNAهای راهنما برای هدف قرار دادن توالیهای غیرپالیندرومیک خاص موجود در موجودات مهاجم استفاده میکنند. آنها میتوانند DNA با طول متغیر را برش دهند، مشروط بر اینکه یک RNA راهنمای مناسب ارائه شود. انعطاف پذیری و سهولت استفاده از این نوع آنزیمها، آنها را برای کاربردهای مهندسی ژنتیک در آینده امیدوار میسازد.
آنزیمهای محدودکننده مصنوعی
آنزیمهای محدودکننده مصنوعی را میتوان با ادغام یک دومین اتصال طبیعی یا مهندسی شده DNA به یک دومین نوکلئاز (اغلب دومین برش از نوع آنزیم محدودکننده IIS FokI) تولید نمود. چنین آنزیمهای محدودکننده مصنوعی میتوانند بخشهای DNA بزرگ (تا ۳۶ جفت باز) را هدف قرار دهند و برای اتصال به توالیهای DNA مورد نظر، مهندسی شوند. نوکلئازهای انگشت روی (Zinc finger nucleases) رایجترین آنزیمهای محدودکننده مصنوعی هستند و عموماً در مهندسی ژنتیک مورد استفاده قرار میگیرند، اما میتوانند برای کاربردهای استاندارد شبیهسازی ژن نیز استفاده شوند. دیگر آنزیمهای محدودکننده مصنوعی بر اساس دومین اتصال DNA از افکتورهای TAL هستند. در سال ۲۰۱۳، فناوری جدید CRISPR-Cas9، بر اساس یک سیستم دفاعی ویروسی پروکاریوتی، برای ویرایش ژنوم مهندسی شد و به سرعت در آزمایشگاهها مورد استفاده قرار گرفت.
در سال ۲۰۱۷، گروهی از دانشگاه ایلینویز گزارش دادند که از پروتئین Argonaute گرفته شده از Pyrococcus furiosus (PfAgo) به همراه DNA راهنما برای ویرایش DNA در شرایط آزمایشگاهی به عنوان آنزیمهای محدودکننده مصنوعی استفاده کردند. ریبونوکلئازهای مصنوعی که به عنوان آنزیمهای محدودکننده RNA عمل میکنند نیز ساخته شدهاند. سیستم مبتنی بر PNA، به نام PNAzyme، دارای یک گروه Cu(II)-2،9-dimethylphenanthroline است که ریبونوکلئازها را برای توالی RNA خاص تقلید میکند و هنگام اتصال آنزیم به RNA، در ناحیه غیر جفت باز (برآمدگی RNA) از RNA هدف را برش میدهد. این آنزیم قدرت انتخاب کنندگی خود را تنها با برش در یک محل که یا تطابق دارد یا از نظر جنبشی (سینتیکی) از بین دو محل برش احتمالی ترجیح داده میشود، نشان میدهد.
نامگذاری
از زمان کشف آنها در دهه ۱۹۷۰، بسیاری از آنزیمهای محدودکننده شناسایی شدهاند. به عنوان مثال، بیش از ۳۵۰۰ آنزیم محدودکننده نوع II مختلف شناسایی شده است. هر آنزیم با استفاده از یک سیستم نامگذاری بر اساس جنس، گونه و سویه باکتری، از روی باکتری که از آن جدا شده است، نامگذاری میگردد. به عنوان مثال، نام آنزیم محدود کننده EcoRI در کادر زیر نشان داده شده است.
| مشتق شده از نام EcoRI | ||
| مخفف | معنی | تشریح |
| E | Escherichia | جنس |
| co | coli | گونههای خاص |
| R | RY13 | گونه |
| I | ابتدا شناسایی شد | ترتیب شناسایی در باکتری |
کاربردها
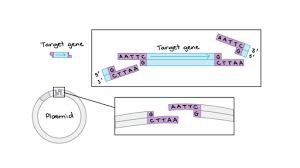
آنزیمهای محدودکننده ایزوله برای تغییر و دستکاری DNA و کاربردهای علمی مختلف استفاده میشوند. آنها برای کمک به قرار دادن ژنها در ناقلهای پلاسمید در طول کلونینگ ژن و آزمایشهای تولید پروتئین استفاده میشوند. برای استفاده بهینه، پلاسمیدهایی که معمولاً در کلونینگ ژن نقش دارند، اصلاح میشوند تا شامل یک توالی پلیلینکر کوتاه (به نام مکان کلونینگ چندگانه یا MCS) غنی از توالیهای شناسایی آنزیم محدودکننده شوند. این موضوع، امکان انعطاف پذیری را هنگام تعبیه کردن قطعات ژن در ناقل پلاسمید فراهم میسازد. مکانهای برش داده شده بهطور طبیعی در ژنها بر انتخاب اندونوکلئاز برای هضم DNA تأثیر میگذارند، زیرا لازم است تا از برش DNA مورد نظر در هنگام برش عمدی انتهای DNA اجتناب گردد. جهت کلونینگ قطعه ژن در یک وکتور، معمولاً DNA پلاسمید و ژن الحاقی هر دو با همان آنزیمهای محدودکننده بریده میشوند و سپس با کمک آنزیمی به نام DNA لیگاز به هم اتصال مییابند. از آنزیمهای محدودکننده نیز میتوان برای متمایز کردن آللهای ژنی با شناسایی تغییرات تک باز در DNA به نام پلیمورفیسمهای تک نوکلئوتیدی (SNPs) استفاده کرد. با این وجود، این تنها در صورتی امکان پذیر است که یک SNP، محل برش موجود در آلل را تغییر دهد. در این روش میتوان از آنزیم محدودکننده برای ژنوتایپ نمونه DNA بدون نیاز به توالی یابی گران قیمت استفاده نمود. نمونه ابتدا با آنزیم محدودکننده برای تولید قطعات DNA برش داده میشود و سپس قطعات حاصل با اندازههای مختلف از طریق ژل الکتروفورز جدا میشوند. به طور کلی، آللهایی که محلهای برش صحیحی دارند، دو باند DNA قابل مشاهده روی ژل ایجاد میکنند و آن دسته که مکانهای برش تغییر یافته دارند، بریده نمیشوند و تنها یک نوار تولید میکنند. نقشه DNA از طریق برش با آنزیمهای محدودکننده نیز میتواند ایجاد شود تا موقعیتهای نسبی ژنها را نشان دهد. طولهای مختلف DNA تولید شده توسط برش نیز پس از الکتروفورز ژل، الگوی خاصی از نوارها را ایجاد میکند و می تواند برای انگشت نگاری DNA استفاده شود.
همچنین در روش مشابهی، از آنزیمهای محدودکننده جهت بریدن DNA ژنومی برای آنالیز ژن توسط ساترن بلات استفاده میگردد. این تکنیک به محققان اجازه میدهد تا تشخیص دهند که چه تعداد نسخه (یا پارالوگ) از یک ژن در ژنوم یک فرد وجود دارد، یا چه تعداد جهش ژنی (پلیمورفیسم) در یک جمعیت رخ داده است. مثال اخیر چند شکلی یا پلیمورفیسم طول قطعه بریده شده (RFLP) نامیده میگردد. آنزیمهای محدودکننده مصنوعی ایجاد شده از طریق اتصال دومین برش DNA FokI با آرایهای از پروتئینهای متصل شونده به DNA یا آرایههای انگشت روی، که به نوکلئازهای انگشت روی (ZFN) نشان داده میشوند، به دلیل ویژگی توالی ارتقا یافته، ابزاری قدرتمند برای ویرایش ژنوم میزبان هستند. ZFN به صورت جفت کار میکند، دایمریزاسیون آنها در محل از طریق دومین FokI انجام میگیرد. هر آرایه انگشت روی (ZFA) قادر به تشخیص ۱۲-۹ جفت باز است که ۲۴-۱۸جفت باز را ایجاد میکند. یک فاصله ۷-۵ جفت بازی بین محلهای برش، ویژگی اساسی ZFN را افزایش میدهد و آنها را به ابزاری ایمن و دقیقتر تبدیل میکند که میتواند در انسان نیز اعمال گردد. کارآزمایی بالینی فاز اول اخیر برای ZFN جهت لغو هدفمند گیرنده CCR5 برای HIV-1 انجام گرفته است.
در مطالعات دیگر، استفاده از سیستم R-M باکتری را به عنوان مدلی برای ابداع ژن ضد ویروسی انسانی یا واکسنها و درمانهای ژنومی پیشنهاد کردهاند، زیرا سیستم RM نقش دفاعی ذاتی در باکتریها را با محدودسازی گرایش توسط باکتریوفاژها ایفا میکند. پژوهشهای انجام گرفته بر روی REases و ZFN یافت میشود که میتواند DNA ویروسهای انسانی مختلف، از جمله HSV-2، HPVs پرخطر و HIV-1 را با هدف نهایی القای جهشزایی هدف و انحراف ویروسهای آلوده به انسان، از بین ببرد. ژنوم انسان در حال حاضر حاوی بقایایی از ژنوم های رتروویروسی است که غیرفعال و مهار شدهاند.
در واقع، به نظر میرسد مکانیسمهای خاموش کردن عناصر فعال ژنومی L1 توسط سه اگزونوکلئاز ترمیم اصلی 1 (TREX1) و تکمیل کننده متقاطع ترمیم برش 1 (ERCC) از عملکرد سیستمهای RM در باکتریها و اتصال انتهایی غیرهمولوگ (NHEJ) تقلید میکنند که به دنبال استفاده از ZFN بدون الگوی تعمیری است.
مثالها
نمونههایی از آنزیمهای محدودکننده عبارتند از:
| آنزیم | منبع | توالی تشخیصی | برش |
| EcoRI | Escherichia coli | 5’GAATTC
3’CTTAAG |
5′—G AATTC—3′
3′—CTTAA G—5′ |
| EcoRII | Escherichia coli | 5’CCWGG
3’GGWCC |
5′— CCWGG—3′
3′—GGWCC —5′ |
| BamHI | Bacillus amyloliquefaciens | 5’GGATCC
3’CCTAGG |
5′—G GATCC—3′
3′—CCTAG G—5′ |
| HindIII | Haemophilus influenzae | 5’AAGCTT
3’TTCGAA |
5′—A AGCTT—3′
3′—TTCGA A—5′ |
| TaqI | Thermus aquaticus | 5’TCGA
3’AGCT |
5′—T CGA—3′
3′—AGC T—5′ |
| NotI | Nocardia otitidis | 5’GCGGCCGC
3’CGCCGGCG |
5′—GC GGCCGC—3′
3′—CGCCGG CG—5′ |
| HinFI | Haemophilus influenzae | 5’GANTC
3’CTNAG |
5′—G ANTC—3′
3′—CTNA G—5′ |
| Sau3AI | Staphylococcus aureus | 5’GATC
3’CTAG |
5′— GATC—3′
3′—CTAG —5′ |
| PvuII* | Proteus vulgaris | 5’CAGCTG
3’GTCGAC |
5′—CAG CTG—3′
3′—GTC GAC—5′ |
| SmaI* | Serratia marcescens | 5’CCCGGG
3’GGGCCC |
5′—CCC GGG—3′
3′—GGG CCC—5′ |
| HaeIII* | Haemophilus aegyptius | 5’GGCC
3’CCGG |
5′—GG CC—3′
3′—CC GG—5′ |
| HgaI[77] | Haemophilus gallinarum | 5’GACGC
3’CTGCG |
5′—NN NN—3′
3′—NN NN—5′ |
| AluI* | Arthrobacter luteus | 5’AGCT
3’TCGA |
5′—AG CT—3′
3′—TC GA—5′ |
| EcoRV* | Escherichia coli | 5’GATATC
3’CTATAG |
5′—GAT ATC—3′
3′—CTA TAG—5′ |
| EcoP15I | Escherichia coli | 5’CAGCAGN25NN
3’GTCGTCN25NN |
5′—CAGCAGN25 NN—3′
3′—GTCGTCN25NN —5′ |
| KpnI[78] | Klebsiella pneumoniae | 5’GGTACC
3’CCATGG |
5′—GGTAC C—3′
3′—C CATGG—5′ |
| PstI[78] | Providencia stuartii | 5’CTGCAG
3’GACGTC |
5′—CTGCA G—3′
3′—G ACGTC—5′ |
| SacI[78] | Streptomyces achromogenes | 5’GAGCTC
3’CTCGAG |
5′—GAGCT C—3′
3′—C TCGAG—5′ |
| SalI[78] | Streptomyces albus | 5’GTCGAC
3’CAGCTG |
5′—G TCGAC—3′
3′—CAGCT G—5′ |
| ScaI*[78] | Streptomyces caespitosus | 5’AGTACT
3’TCATGA |
5′—AGT ACT—3′
3′—TCA TGA—5′ |
| SpeI | Sphaerotilus natans | 5’ACTAGT
3’TGATCA |
5′—A CTAGT—3′
3′—TGATC A—5′ |
| SphI[78] | Streptomyces phaeochromogenes | 5’GCATGC
3’CGTACG |
5′—GCATG C—3′
3′—C GTACG—5′ |
| StuI*[79][80] | Streptomyces tubercidicus | 5’AGGCCT
3’TCCGGA |
5′—AGG CCT—3′
3′—TCC GGA—5′ |
| XbaI[78] | Xanthomonas badrii | 5’TCTAGA
3’AGATCT |
5′—T CTAGA—3′
3′—AGATC T—5′ |
خلاصه
آنزیمهای محدودکننده، اندونوکلئاز محدودکننده یا به اصطلاح محدودکنندهها، آنزیمهایی هستند که DNA را به قطعاتی در داخل یا نزدیک مکانهای شناسایی خاص درون مولکولهایی که به عنوان مکانهای محدود شده شناخته میشوند، میشکنند. اندونوکلئازهای محدودکننده طبیعی بر اساس ترکیب و نیازهای کوفاکتور آنزیمی، ماهیت توالی هدف و موقعیت محل برش DNA آنها نسبت به توالی هدف به چهار گروه (نوع I، II III و IV) دسته بندی میشوند.
نام مترجم: نگارالسادات موسوی
مطالعات بیشتر در بخش راهنمای علمی سایت




چطوری آنزیمهای محدودگر DNA فاژ را مسدود میکنند ولی به DNA کروموزومی باکتری کاری ندارند
با سلام DNA کروموزومی باکتری در نواحی شناسایی آنزیم (Restriction sites) متیله می باشد