دستهبندی نشده
آنالیز غنیسازی با enrichr، David و GSEA و ژن انتولوژی

درباره آنالیز غنیسازی
آنالیز غنیسازی یا Enrichment Analysis مسیری است که به محققان کمک میکند تا بینش مکانیستیکی درباره فهرستهای ژنی که از قبل تولید کردهاند به دست آورند. این فهرست ژنی شامل ژنهای هدف ماست که در طی فرایندهای قبلی تصمیم گرفتهایم تحلیلهای خود را بر روی آنها انجام دهیم تا بیشتر با عملکرد این ژنها در سلول آشنا شویم.
روش انریچمنت آنالیز برای pathway، در حقیقت مسیرهای بیولوژیکی را شناسایی میکند که در فهرست ژنی بیش از آنچه که به طور تصادفی انتظار میرود غنی شدهاند. مثلاً در نظر بگیرید دستهای ژن دارید. مسلماً ژنهای بیشتری در دیتابیسها وجود دارد، اما شما میخواهید چند ژنی که دارید را بررسی کنید و ببینید که آیا ارتباطی با هم دارند یا خیر.
زمانی که شما این دسته ژن را با این روش تحلیل میکنید الگوریتم در اصل پاسخ این دسته ژنهای شما را با حالات تصادفی مقایسه میکند. یعنی میگوید اگر یک دسته رندوم ژن بردارم و این تحلیل را انجام دهم، چقدر احتمال دارد این ژنها با هم در ارتباط باشند. مسلماً یک دسته ژن رندوم، نباید ارتباط خاصی را نشان دهند و امتیاز آماری آن پایین خواهد بود.
پس اگر پاسخ شما نشان میدهد که ژنها یک عملکرد خاص دارند و در یک مسیر فعالیت میکنند، این نتیجه از لحاظ آماری نیز باید امتیاز خوبی را کسب کند تا شما با اطمینان بیشتری بگویید دسته ژنهایی که داشتهاید واقعاً یک رابطه معناداری با هم دارند و تصادفی انتخاب نشدهاند.
پروتکل آنالیز غنیسازی دسته ژن یا مسیر را میتوان در سه مرحله خلاصه کرد در طی آن شما در ابتدا فهرستی از ژنهای دخیل در بیماری را به دست میآورید. این ژنها با کمک دادههای اومیکس به دست آمدهاند. در مرحله بعدی شما باید به کمک الگوریتمهای انریچمنت آنالیز دسته ژن، بهصورت آماری دادههای خود را بررسی کنید. در مرحله آخر نیز نتایج را باید مصور کنید تا بتوانید آن را تفسیر کنید.
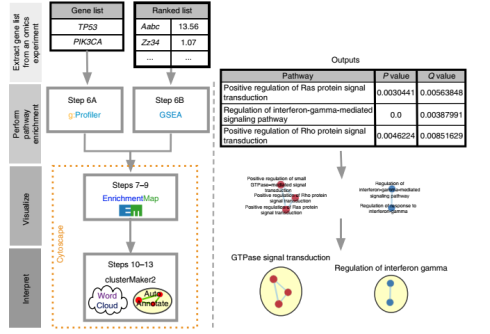
نرمافزارهای مختلفی برای این سه قدم کار آماده شدهاند و بهصورت رایگان در دسترس شما هستند. از جمله این نرمافزارها میتوان به g:Profiler، Gene Set Enrichment Analysis (GSEA)، Cytoscape و EnrichmentMap اشاره کرد.
در این مسیر برای تحلیل، شما به کمک نرمافزارهایی مثل R یا سرور گلکسی تحلیلهای آماری را انجام داده و لیستی از ژنهای اولیه را به دست میآورید. سپس این بر روی این لیست ژنها باید به کمک پلتفرمهایی مثل g:profiler و GSEA ژن انتولوژی و آنالیز غنیسازی انجام شود. نتیجه حاصل نیز میتواند در نرمافزار سایتواسکیپ وارد شده و مصورسازی میشود. همچنین این نتایج به کمک سایتواسکیپ میتواند از لحاظ شبکه نیز تحلیل شود. این نرمافزار خود اپلیکیشنهایی دارد که به کمک آنها میتوانید دستورات مختلفی را اجرا کنید و از Webstore نیز دسترسی دارد. یکی از این موارد EnrichmentMap است.
شما به کمک لینکهای زیر میتوانید به این پلتفرمها دسترسی داشته باشید:
- g:Profiler (https://biit.cs.ut.ee/gprofiler/)
- GSEA (http://software.broadinstitute.org/gsea/)
- Cytoscape (http://www.cytoscape.org/)
- EnrichmentMap (http://www.baderlab.org/Software/EnrichmentMap)
ابزارهای مختلفی برای انجام GSEA یا آنالیز غنیسازی دسته ژن وجود دارد. GSEA از روشهای آماری مختلفی برای انریچمنت آنالیز استفاده میکند به همین دلیل برای اجرای این محاسبات به کامپیوتر احتیاج دارد. این روش استاندارد بوده و سایتها و نرمافزارهای مختلفی هستند که از آن برای تحلیل مجموعهدادهها استفاده میکنند. در زیر به تعدادی از این وبسایتها اشاره شده است:
MSigDB و Broad Institute
Molecular Signatures Database مجموعه گستردهای از ژنها دارد که میتواند در بیشتر نرمافزارهای GSEA استفاده شود. Broad Institute نیز به این دیتابیس متصل بوده و خود یک نرمافزار برای تحلیل GSEA بهصورت آنلاین در اختیار دارد.
Enrichr
Enrichr یک ابزار معروف تحلیل غنیسازی دستهای از ژنهای دخیل در بیماری در پستانداران است. این ابزار کتابخانههای مختلفی دارد که شامل موارد زیر میشود:
- کتابخانههایی برای تنظیم رونویسی
- کتابخانههای مسیرهای زیستی و تعاملات پروتئینی
- هستیشناسی ژن
- سلولهای تیمارشده با دارو
- بیان ژنها در سلولها و بافتهای مختلف
از دیگر وبسایتها و پایگاهدادههای مهم که مورداستفاده قرار میگیرند نیز میتوان به موارد زیر اشاره کرد:
- DAVID
- g:Profiler
- Metascape
- Genomic region enrichment of annotations tool (GREAT)
- PlantRegMap: در حوزه گیاهی استفاده میشود.
هستیشناسی ژنی
حاشیهنویسی ژنی یا annotation بر اساس هستیشناسی ژنی یا همان Gene Ontology به ما کمک میکند که یک ساختار سلسلهمراتبی را از آنچه درون سلول رخ میدهد داشته باشیم. در حقیقت این اطلاعات مانند یک لغتنامه برای ما کار میکند که میتواند درک بهتری از اجزای سلول، فرایندها و مکان آنها به ما بدهد.

تلاش شده است که اطلاعات درون این لغتنامه منحصربهفرد باشد و همچنین به کمک آن بتوانیم ارتباط بین اجزا را شناسایی کنیم. به دلیل آنکه چندین اسم برای یک مفهوم وجود داشت و یا اینکه یک اسم به چندین مفهوم اطلاق میشد، شاهد نوعی بیثباتی یا inconsistency در زبان بودیم. مشکلی که درک مفاهیم را برای ما سخت میکرد.
درنتیجه، هستیشناسی ژنی یا ژن انتولوژی قصد داشته که چنین مشکلی را حل کند و به همین دلیل ایجاد شده است. مثلاً بعضی از محققین بهجای استفاده از سنتز گلوکز از عبارت گلوکز بیوسنتز، گلوکز فرمیشن، گلوکز آنابولیسم یا گلوکونئوجنسیس استفاده میکردند که همگی به تولید زیست اشاره میکند. اینگونه بیثباتیها سبب شد که یک لغتنامه کلی برای این مشکل ایجاد شود تا همگی بر روی نام استفاده شده اتفاقنظر داشته باشد و به یک اسم یا مفهوم جامع تبدیل شود.
کاربرد دیگری که ژن انتولوژی یا هستیشناسی دارد این است که به کمک آن میتوانیم مجموعهدادههای بزرگ را بررسی کنیم. کاری که برای بررسی انجام میدادیم خوشهبندی بود. به کمک الگوریتمهای بیوانفورماتیک میتوانیم عملکردی را به این ژنهای مشابه اختصاص دهیم تا عملکرد را بررسی کنیم.
با وجود توسعه روشهای اومیکس، اطلاعات در سطوح مختلف به دست آمده است که باید annotation برای آنها نیز انجام شود. به همین دلیل نیاز به یک روش annotation بهصورت اتوماتیک داریم که بتواند این کار را بهخوبی و با سرعت بیشتری انجام دهد.
علاوه بر مشکلاتی که درمورد اسمها وجود داشت، بین ارگانیسمهای متفاوت نیز تفاوت وجود دارد. مثلاً در بین ارگانیسمها، به یک پروتئین مشابه نامهای مختلفی اطلاق میشد که باز این موضوع نیز بر پیچیدگی ماجرا اضافه میکرد. هستیشناسی ژن یا ژن انتولوژی قصد داشته که این مشکل را نیز برطرف کند.
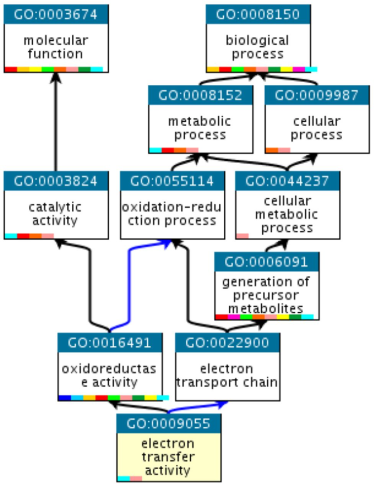
این حوزه به سه دسته کلی تقسیمبندی میشود. ما برای ژنها و محصولات پاییندستی آنها سه دسته اطلاعات به دست میآوریم که دسته اول میگوید در چه فرایندی دخیل است. دسته دوم اشاره میکند که این ژن از لحاظ مولکولی دقیقاً چه عملکردی را انجام میدهد، مثلاً محصول ژن به چه چیزی متصل میشود و اینکه چه کاری میکند. در دسته سوم نیز درمورد جایگاه آن در سلول صحبت میشود که میگوید مثلاً پروتئین در غشا قرار دارد یا در سیتوزول. درنتیجه ما در ژن انتولوژی سه دسته اطلاعات خواهیم داشت و یک ترم یونیک برای هرکدام از اجزا استفاده میشود.
فرایند بیولوژیکی (biological process) به یک هدف بیولوژیکی اشاره دارد که ژن یا محصول ژنی به ایجاد آن کمک میکند. یک فرایند از طریق یک یا چند مجموعه منظم از توابع مولکولی انجام میشود. فرایندها اغلب شامل یک دگرگونی شیمیایی یا فیزیکی هستند، به این معنا که چیزی وارد یک فرایند میشود و چیز متفاوتی از آن خارج میشود.
نمونههایی از اصطلاحات فرایند بیولوژیکی (فرایندهای سطح بالا) عبارتاند از “رشد و نگهداری سلول” (cell growth and maintenance) یا “تبدیل سیگنال” (signal transduction). نمونههایی از اصطلاحات فرایندی خاصتر (فرایندهای سطح پایینتر) عبارتاند از «ترجمه» (translation)، «متابولیسم پیریمیدین» (pyrimidine metabolism) یا «بیوسنتز cAMP» (cAMP biosynthesis).
عملکرد مولکولی (molecular function) بهعنوان فعالیت بیوشیمیایی (شامل اتصال خاص به لیگاندها یا ساختارها) در یک محصول ژنی تعریف میشود. این سطح فقط آنچه را که انجام میشود بدون مشخصکردن مکان یا زمان وقوع رویداد توصیف میکند. نمونههایی از اصطلاحات کاربردی گسترده عبارتاند از «آنزیم»، «حامل» یا «لیگاند». نمونههایی از اصطلاحات کاربردی خاصتر عبارتاند از “آدنیلات سیکلاز” یا “لیگاند گیرنده Toll”.
جزء سلولی (cellular component) به مکانی در سلول اشاره دارد که یک محصول ژن در آن فعال است. این اصطلاحات درک ما از ساختار سلول یوکاریوتی را نشان میدهد. همانطور که برای دیگر هستیشناسیها صادق است، همه اصطلاحات برای همه موجودات قابلاستفاده نیستند. مؤلفه سلولی شامل عباراتی مانند «ریبوزوم» یا «پروتئزوم» است که مشخص میکند محصولات چند ژن در کجا یافت میشوند. همچنین این اصلاحات شامل عباراتی مانند “غشای هستهای” یا “دستگاه گلژی” نیز میشوند.
ویژگی دیگر ژن انتولوژی یا هستیشناسی ژن، وجود ارتباط بین این اجزای سلولی است. فرایندها میتوانند زیرمجموعه هم قرار بگیرند و به همین دلیل یک ساختار سلسلهمراتبی بین آنها وجود دارد. مثلاً biological process به دودسته metabolic process و cellular process تقسیم میشود و هرکدام از این موارد بهمراتب ریزتر تقسیم میشوند. گرافی که در اینجا داریم جهتدار است و نودهای گراف را بهصورت سلسلهمراتبی به هم متصل میکند. نکته دیگر اینکه گراف هیچ دوری ندارد و تنها در یک جهت حرکت میکند.
دوره کارآموزی طراحی و مدلسازی سلولی: سیستم بیولوژی
منبع:
Ashburner M, Ball CA, Blake JA, et al. Gene ontology: tool for the unification of biology. The Gene Ontology Consortium. Nat Genet. 2000;25(1):25-29. doi:10.1038/75556
Reimand, J., Isserlin, R., Voisin, V. et al. Pathway enrichment analysis and visualization of omics data using g:Profiler, GSEA, Cytoscape and EnrichmentMap. Nat Protoc 14, 482–517 (2019). https://doi.org/10.1038/s41596-018-0103-9



