دستهبندی نشده
آشنایی با Cytoscape ، پلاگینهای معروف و کار با آنها
درباره نرمافزار سایتواسکیپ (Cytoscape)
نرمافزار سایتواسکیپ (Cytoscape) یک نرمافزار اوپن سورس است، یعنی نرمافزاری که برای استفاده از آن نیازی به پرداخت پول نیست که قصد یکپارچهسازی شبکههای ارتباطات بیوموملکولها که با روشهای دارای توان بالا به دست آمدهاند را دارد. اگرچه سیتواسکیپ برای هر سیستمی از اجزای مولکولی و برهمکنشها قابلاستفاده است، اما سایتواسکیپ زمانی قویتر عمل میکند که همراه با پایگاههای داده بزرگ پروتئین – پروتئین، پروتئین -DNA، و برهمکنشهای ژنتیکی که برای انسانها و ارگانیسمهای مدل در دسترس هستند، استفاده شود.
این نرمافزار قابلیتهای زیادی برای تحلیل شبکههای زیستی و غیرزیستی دارد و دانشگاهها و موسسات تحقیقاتی بزرگی در سرتاسر دنیا درحال توسعه پلاگینهایی برای این نرمافزار هستند. پلاگینها در حقیقت پکیجهایی هستند که به تحلیل شبکه کمک میکنند. مثلا شما میتوانید به کمک پلاگین GeneMania، شبکهای از دستهای از ژنهای انتخاب شده را به کمک دیتابیس این پلاگین رسم کنید و از لحاظ GO بررسی کنید. ببینید که هر ژن در چه مسیری قرار دارد و در این مسیر زیستی با دیگر ژنهای انتخاب شده ارتباط دارد یا نه.
علاوه بر بحث پلاگینها، شما میتوانید به کمک خود سایتواسکیپ نیز شبکه را ترسیم کنید، نحوه نمایش گرهها و یالها را تغییر دهید و آنها را بر حسب دیتای ورودی تغییر دهید. مثلا اگر ارتباط بین دستههای ژنی حاصل از نرمافزار GESA را رسم میکنید، میتوانید بر حسب تعداد ژنها اندازه گرهها را تغییر دهید. یعنی هرچقدر که ژن بیشتری در یک دسته ژنی بود، گره بزرگتر رسم شود. ضخامت یال را نیز میتوانید به همین شکل تعیین کنید. مثلا هرچقدر تعداد ژنهای یکسانی در دو دسته ژنی وجود داشت، یال ضخامت بیشتری بگیرد.
در حقیقت به کمک سایتواسکیپ، شما میتوانید شبکههایی را رسم کرده و تحلیل کنید که به شما کمک میکنند دید بهتری از سوال زیستی خود داشته باشید. مثلا با رنگآمیزی نودها و یالها برحسب امتیاز adjusted p value، شما میتوانید اهمیت گرهها و یالها را با یک نگاه تشخیص دهید.
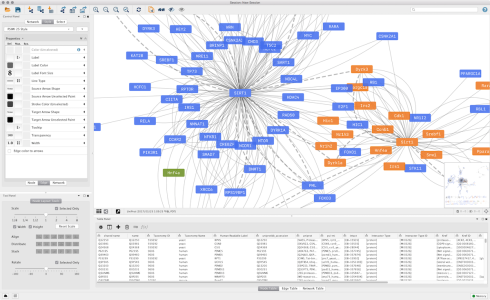
البته گاهی اوقات آن قدر شبکه پیچیده است که این رویکرد مناسب نیست. باز شما میتوانید به کمک بخش filter که در این نرمافزار وجود دارد، شبکه خود را برحسب ویژگیهایی که تعیین میکنید کوچکتر کنید، تا به بخش مهم و اصلی آن برسید و آن را گزارش کنید. به کمک بخش آنالیز نتورک نیز، سایتواسکیپ اطلاعات کلی درمورد توپولوژی نتورک را در اختیار شما قرار میدهد. در شکل زیر تصویری از این سایت را مشاهده میکند.
در بخش پایین صفحه، شما اطلاعات دیتاست را مشاهده میکنید که حاوی اطلاعاتی از نود و یال است. در این بخش مشخص میشود که جنس هر یال چیست. مثلا اگر شما یک شبکه ارتباط پروتئین داشته باشید، جنس یالهای شما میتواند ارتباط فیزیکی تایید شده در آزمایشگاه، روشهای insilico مانند text mining یا بیان ژنی باشد. تحلیل اطلاعات مختلف به صورت همزمان تنها به کمک این دست از ابزار سیستم بیولوژی ممکن خواهد بود و با بررسیهای ساده نمیتوان دادههای مختلف را کنار هم بررسی کرد.
سایتواسکیپ بر اساس جاوا نوشته شده است و برای آنکه بتوانید بهراحتی از آن استفاده کنید باید این پلتفرم را بر روی کامپیوتر خود داشته باشید و آن را آپدیت کنید. سایتواسکیپ همواره در حال پیشرفت بوده و نسخههای جدید آن همواره ارائه میشوند. از ماه می 2016، انتقال از Cytoscape 2.x به 3.x کامل شد. امروزه سایتواسکیپ 3 نسخه اصلی سایتواسکیپ است و از نسخه دو دیگر استفاده نمیشود و به همین دلیل این شرکت این نسخه را پشتیبانی نمیکند. بااینحال، اگر قصد دارید که از پلاگینهای این نسخه استفاده کنید میتوانید ورژن Cytoscape 2.8.3 استفاده کنید، اما در غیر این صورت، لطفاً از آخرین نسخه سری Cytoscape 3.x استفاده کنید.
برای افزایش قابلیتهای این نرمافزار پرقدرت، میتوانید از پلاگینهای مختلف آن استفاده کنید که بهصورت رایگان در سایت سایتواسکیپ قرار میگیرد. بسته به نیازی که دارید میتوانید هر پلاگینی را روی core برنامه نصب کنید تا از قابلیتهای آن استفاده کنید. در زیر نام تعدادی از پرکاربردترین پلاگینهای مورداستفاده در سایتواسکیپ آورده شده است. پلاگینها نیز در طول زمان توسعه پیدا میکنند و ورژنهای جدید آنها در App store cytoscape قابلدسترسی است.
GeneMANIA
این پلاگین، شبکههای تعاملی را از پایگاههای داده از فهرستی از ژنها همراه با حاشیهنویسیها و عملکردهای آنها را وارد برنامه میکند.
پلاگین GeneMANIA قابلیتهای پیشبینی عملکرد ژن را به شما میدهد. GeneMANIA مرتبطترین ژنها را با یک مجموعه ژن کوئری با استفاده از رویکرد guilt-by-association شناسایی میکند. این افزونه از یک پایگاهداده بزرگ از شبکههای تعامل عملکردی از ارگانیسمهای متعدد استفاده میکند. کاربران ممکن است شبکههای تعاملی و دادههای بیان خود را برای تکمیل یا نادیدهگرفتن دادههای پیشفرض اضافه کنند.
BiNGO
BiNGO ابزاری است برای تعیین اینکه کدام دستهبندیهای هستیشناسی ژن (GO) از نظر آماری به طور قابلتوجهی در مجموعهای از ژنها یا زیرگراف یک شبکه بیولوژیکی نشان داده شدهاند. BiNGO مضامین عملکردی غالب یک مجموعه ژن معین را بر اساس سلسلهمراتب GO ترسیم میکند و این نگاشت را بهعنوان یک گراف سایتواسکیپ خروجی میدهد.
مجموعههای ژنی را میتوان از یک شبکه Cytoscape (بهعنوان زیرگراف) انتخاب یا محاسبه کرد یا از منابعی غیر از Cytoscape (مثلاً فهرستی از ژنهایی که در یک آزمایش میکرواری به طور قابلتوجهی بیان داشتهاند) جمعآوری کرد.
بینگو میتواند به شما در مصورسازی دادههایی که در اختیار دارید کمک کند. امکانات این برنامه عبارتاند از:
- ارزیابی over-representation یا under-representation از دستههای GO
- لیست ژن یا لیست گراف در ورودی
- حالت دستهای (batch mode): میتوانید با استفاده از تنظیمات یکسان، چندین خوشه را به طور همزمان تجزیهوتحلیل کنید.
- هستیشناسیهای مختلف GO و GOSlim
- استفاده از طیف گستردهای از ارگانیسمها
- تصحیح تست چندگانه با استفاده از اصلاح Bonferroni (FWER) یا Benjamini&Hochberg (FDR)
- تجسم تعاملی نتایج نگاشتشده در سلسلهمراتب GO
- این پلاگین متنباز بوده و بهراحتی میتوانید از آن استفاده کنید.
MCODE
MCODE یک شبکه داده شده را بر اساس توپولوژی برای یافتن مناطق متراکم خوشهبندی میکند. MCODE خوشهها (مناطق بسیار بههمپیوسته) را در یک شبکه پیدا میکند. خوشهها در انواع مختلف شبکهها معانی مختلفی دارند. بهعنوانمثال، خوشهها در یک شبکه تعامل پروتئین – پروتئین (protein-protein interaction network) اغلب مجتمعهای پروتئینی و بخشهایی از مسیرها هستند، درحالیکه خوشهها در شبکه شباهت پروتئین نشاندهنده (protein similarity network) خانوادههای پروتئینی هستند.
AgilentLiteratureSearch
مقالات را برای یافتن نشریات مرتبط با عبارات جستجوشده بررسی میکند و شبکههای تعاملی بر اساس نتایج جستجو را ایجاد میکند. این پلاگین، یک ابزار فراجستجو برای جستجوی خودکار چندین موتور جستجوی مبتنی بر متن (اعم از عمومی و اختصاصی) است تا به زیستشناسان کمک کند و دیگر نیازی به جستوجوی دستی برای پیداکردن چنین نتایجی نیست.
این نرمافزار را میتوان همراه با Cytoscape استفاده کرد که ابزاری برای ایجاد نمای شبکهای کلی از ارتباطهای ژن-پروتئین ارائه میدهد. همچنین این نرمافزار یک رابط کاربری آسان برای قابلیتهای جستجوی قدرتمند فراهم میکند. هنگامی که یک کوئری وارد میشود، به چندین موتور جستجوی انتخاب شده توسط کاربر ارسال شده و نتایج بازیابی شده از منابع مربوطه دریافت میشود. سپس هر نتیجه به اجزایش تجزیه میشود و برای ارتباط پروتئین – پروتئین تحلیل میشود.
از دیگر پلاگینهای پرکاربرد در سایتواسکیپ میتوان به ClueGO، CluePedia، cyREST، stringApp، yFiles Layout Algorithms و غیره اشاره کرد که هرکدام کاربردها و قابلیتهای مخصوص خود را برای تحلیل شبکه و ساختن شبکههای مختلف دارند. داشتن توانایی کار با این نرمافزار قدرتمند به ما کمک میکند تحلیلهای بهتری از سیستم ارائه دهیم و نمودارهای مدنظر خود را ترسیم کنیم.
سایتواسکیپ با نرمافزارها و پلتفرمهای مختلف دیگری در ارتباط است و شما میتوانید بهراحتی دیتا بین آنها جابهجا کنید. مثلا برای تحلیل شبکهای که در سایت STRING ساختهاید میتوانید با زدن بخش انتقال به سایتواسکیپ در بخش export، شبکه را به محیط سایتواسکیپ منتقل کنید. همچنین به کمک پلاگین EnrichmentAnalysis میتوانید خروجی GSEA را وارد cytoscape کرده تا آن را رسم کنید.
دوره کارآموزی طراحی و مدلسازی سلولی: سیستم بیولوژی
منبع:
Saito R, Smoot ME, Ono K, et al. A travel guide to Cytoscape plugins. Nat Methods. 2012;9(11):1069-1076. doi:10.1038/nmeth.2212
Shannon P, Markiel A, Ozier O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Res. 2003;13(11):2498-2504. doi:10.1101/gr.1239303




